Clasificación de cáncer de mama en imágenes histológicas
Clasificación de carcinoma ductal invasivo usando técnicas de deep learning.
- Objetivos
- Descripción del dataset
- Análisis exploratorio
- Procesamiento de los datos
- Entrenamiento del modelo
- Análisis de los resultados
- Referencias
El cáncer de mama es uno de los tipos de cáncer más frecuentes del mundo. En el año 2019, se diagnosticaron en España 33.307 nuevos casos. Además, entre el año 2012 y 2019 ha habido un incremento de casos del 7,5 %. Aunque la mortalidad de este tipo de cáncer se ha reducido en los últimos años, los casos van a seguir aumentando y los tratamientos siguen siendo agresivos. Por tanto, es necesario contar con mecanismos de diagnóstico temprano para evitar estos tratamientos, y de paso ayudar al personal sanitario con el diagnóstico.
Aquí es donde entra el deep learning. Gracias a los avances de los últimos años, los modelos de aprendizaje profundo pueden diagnosticar enfermedades casi tan bien como los médicos. A lo largo de este post, desarrollaremos un modelo de clasificación de carcinoma ductal invasivo (IDC en inglés), el cáncer de mama más frecuente. Para ello, plantearemos los siguientes objetivos:
Objetivos
- Desarrollar un modelo de clasificación de IDC utilizando la arquitectura VGGNet en Keras.
- Realizar el análisis exploratorio de los datos.
- Aplicar transferencia de aprendizaje y data augmentation.
Descripción del dataset
El dataset que vamos a usar ha sido extraído de la plataforma Kaggle. El dataset está formado por un conjunto de imágenes histológicas tomadas a 279 mujeres. En cada imagen se ha señalado la región más afectada por un experto. Por razones de tamaño y eficiencia durante el entrenamiento, se han dividido las imágenes histológicas en imágenes más pequeñas, etiquetando cada una de ellas con la etiqueta 0 (sin IDC) o 1 (con IDC).
Análisis exploratorio
El primer paso a la hora de trabajar con un conjunto de datos consiste en realizar una exploración para ver la distribución de los datos y si son correctos. Si observamos el dataset, vemos que tenemos un total de 277.524 imágenes con una resolución de 50x50 píxeles. Podemos ver que disponemos de muchísimas imágenes.
A continuación, comprobaremos si existe desbalanceo de clases observando el siguiente histograma:
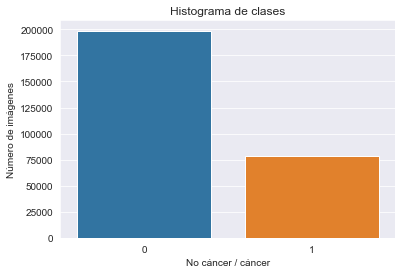
En la imagen anterior vemos que existe un mayor número de imágenes etiquetas sin IDC. Este es un comportamiento frecuente a la hora de analizar datos de imágenes médicas. Por tanto, las clases están desbalanceadas. Si usamos datos desbalanceados, el modelo no conseguirá aprender correctamente las características que definen a una imagen con IDC, ya que el número de imágenes es menor.
Otro de los aspectos a señalar es que las imágenes no son todas del mismo tamaño. Existen varias imágenes con una resolución distinta a 50x50. Estas imágenes pertenecen a los bordes de la imagen histológica y no contienen información relevante para el problema.
Procesamiento de los datos
Con respecto a la gestión del dataset, se han eliminado las imágenes con resolución distinta a 50x50. Por razones de eficiencia y desbalanceo de clases, se ha reducido la muestra a unas 20.000 imágenes balanceadas. A continuación, dividimos la muestra en los conjuntos de entrenamiento, validación y prueba. Para gestionar el gran número de imágenes, he creado una base de datos HDF5 usando el paquete h5py.
| Entrenamiento | Validación | Prueba |
|---|---|---|
| 14.470 imágenes | 2500 imágenes | 3000 imágenes |
A la hora de entrenar el modelo, no debemos cargas tantas imágenes en memoria, debido a la limitación de recursos. Para ello, crearemos un generador en Keras que vaya cargando en memoria las imágenes a medida que se necesiten. También sería interesante que el generador aplique las técnicas de preprocesamiento en tiempo de entrenamiento.
# Load packages
from keras.utils import np_utils
import numpy as np
import h5py
class KerasGenerator:
def __init__(self, db_path, batch_size, preprocessors=[],
data_augmentation=None):
self.preprocessors = preprocessors
self.batch_size = batch_size
self.db = h5py.File(db_path, "r")
self.num_images = self.db["images"].shape[0]
self.data_augmentation = data_augmentation
def generate_image_batch(self):
while True:
for i in np.arange(0, self.num_images, self.batch_size):
images = self.db["images"][i: i + self.batch_size]
labels = self.db["labels"][i: i + self.batch_size]
# Apply preprocessors to images on batch
if [] not in self.preprocessors:
images_preprocessed = []
for image in images:
for preprocessor in self.preprocessors:
image = preprocessor.preprocess(image)
images_preprocessed.append(image)
images = np.array(images_preprocessed)
# Categorizing labels
labels_categorized = []
for label in labels:
label = np_utils.to_categorical(label, num_classes=2)
labels_categorized.append(label)
labels = np.array(labels_categorized)
# Apply data augmentation (optional)
if self.data_augmentation is not None:
(images, labels) = next(self.data_augmentation.flow(images,
labels,
batch_size=self.batch_size))
yield (images, labels)
Para implementar el generador, creamos una clase llamada KerasGenerator. El constructor de la clase recibe como parámetros la ruta de la base de datos, el tamaño de batch, el preprocesamiento a aplicar y si vamos a aplicar data augmentation o no. El método generate_image_batch contiene toda la lógica necesaria para el generador. Se encarga de cargar en memoria el siguiente batch y aplicar los preprocesamientos de la lista self.preprocessors. A continuación, se categorizan las etiquetas para que puedan ser aceptadas por Keras. Finalmente, se aplican las técnicas de aumento de datos, si las hubiera.
Entrenamiento del modelo
Una vez que tenemos los datos preparados, podemos empezar a crear nuestro modelo de deep learning. En primer lugar, importamos los paquetes necesarios:
from keras.applications import VGG16
from keras.optimizers import SGD
from utilities.fclayer import FCLayer
from keras.layers import Input
from keras.models import Model
from utilities.kerasgenerator import KerasGenerator
from keras.preprocessing.image import ImageDataGenerator
import config.breast_histopathology_cancer_config as config
import utilities.preprocessing as preprocessors
import matplotlib.pylab as plt
import numpy as np
import json
A continuación, cargamos los valores de la media RGB del dataset a partir del archivo JSON generado por el script build_breast_histopathology_cancer.py. Este tipo de normalización ofrece mejores resultados que dividir los píxeles por 255.
values_rgb = json.loads(open(config.MEAN_PATH).read())
Definimos las transformaciones de data augmentation que vamos a aplicar durante el entrenamiento. Con data augmentation, la red verá imágenes distintas en cada época, reduciendo el sobreajuste. Las transformaciones serán las siguientes: volteo horizontal y vertical, giros aleatorios de 90º y zoom.
batch_size = 32
number_epochs = 20
aug = ImageDataGenerator(horizontal_flip=True,
vertical_flip=True,
rotation_range=90,
zoom_range=[0.5, 1.0]
)
Definimos el preprocesamiento y los generadores de validación y prueba. Básicamente, redimensionamos las imágenes a 224x224 para aplicar transferencia de aprendizaje y restamos la media RGB a cada una de ellas.
rp = preprocessors.ResizePreprocessor(224, 224)
mrgb = preprocessors.MeanRGBPreprocessor(values_rgb)
trainGen = KerasGenerator(config.TRAIN_HDF5, batch_size,
preprocessors=[rp, mrgb], data_augmentation=aug)
valGen = KerasGenerator(config.VAL_HDF5, batch_size, preprocessors=[rp, mrgb]
A continuación, definimos el modelo a usar, VGG16, con los pesos de ImageNet para aplicar transferencia de aprendizaje. Con esto aprovechamos las características aprendidas en redes preentrenadas y no necesitamos volver a entrenarlas desde cero. Eliminamos la capa totalmente conectada y la sustituimos por otra más sencilla con dropout.
baseModel = VGG16(weights="imagenet", include_top=False,
input_tensor=Input(shape=(224, 224, 3)))
head = FCLayer.build(baseModel, 256)
model = Model(inputs=baseModel.input, outputs=head)
El entrenamiento se llevará a cabo en dos fases, aplicando fine-tuning. En la primera fase congelamos las capas convolucionales de la red y entrenamos solamente la última capa. En la segunda fase, una vez aprendidas las características, descongelamos algunas capas convolucionales y reducimos la tasa de aprendizaje para no cambiar demasiado los pesos. Usamos como optimizador SGD y la entropía cruzada como función de error. El dataset se ha entrenado con la versión GPU de TensorFlow en un ordenador con 16 GB de RAM.
opt = SGD(lr=0.01)
model.compile(loss="categorical_crossentropy", optimizer=opt,
metrics=["accuracy"])
# %% Training model
print("[INFO] Training Model")
H1 = model.fit(trainGen.generate_image_batch(),
steps_per_epoch=trainGen.num_images // batch_size,
validation_data=valGen.generate_image_batch(),
validation_steps=valGen.num_images // batch_size,
epochs=number_epochs,
max_queue_size=batch_size * 2,
verbose=1
)
# %% Unfreeze some CONV layers for learn more features
for layer in baseModel.layers[15:]:
layer.trainable = True
opt = SGD(lr=0.001)
model.compile(loss="categorical_crossentropy", optimizer=opt,
metrics=["accuracy"])
print("[INFO] Training Model")
H2 = model.fit(trainGen.generate_image_batch(),
steps_per_epoch=trainGen.num_images // batch_size,
validation_data=valGen.generate_image_batch(),
validation_steps=valGen.num_images // batch_size,
epochs=number_epochs,
max_queue_size=batch_size * 2,
verbose=1
)
Análisis de los resultados
Una vez acabado el entrenamiento, ya podemos evaluar el modelo en nuestro conjunto de prueba.
| Modelo | VGG16 | Cruz-Roa et. al [1] |
|---|---|---|
| Accuracy | 0.87 | |
| BAC | 0.88 | 0.84 |
Para evaluar el modelo, se han usado dos métricas: accuracy debido a que hemos entrenado un dataset balanceado, y balanced accuracy (BAC), para comparar con el artículo de Cruz-Roa [1]. Podemos ver que aplicando dos técnicas como la transferencia de aprendizaje y data augmentation se consigue mejorar los resultados de clasificación. En Cruz-Roa utilizan una arquitectura convolucional de tres capas sobre el dataset completo, entrenándola desde cero. También se compara esta arquitectura con métodos manuales de extracción de características, que ofrecen peores resultados que las técnicas de deep learning utilizadas.
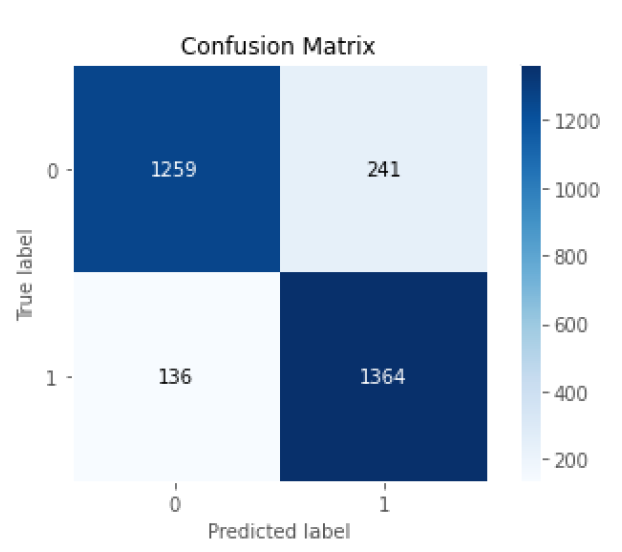
Si observamos la matriz de confusión podemos ver que el modelo consigue clasificar la mayoría de imágenes correctamente. También vemos que tenemos un número muy bajo de falsos negativos. En el campo del diagnóstico de enfermedades, se prefiere minimizar al máximo los falsos negativos en lugar de los falsos positivos, ya que corremos el riesgo de no detectar las zonas afectadas de la enfermedad.
Por último, si juntamos todas las imágenes creando la imagen histológica completa (script probabilitymap.py), podremos ver mejor el rendimiento del modelo.

A la izquierda tenemos las predicciones reales, señaladas por un experto. A la derecha tenemos las predicciones del modelo. Vemos que obtenemos resultados muy similares a los del experto.
A lo largo del post hemos visto como las técnicas de deep learning consiguen resolver problemas complejos de forma precisa. Para ver más detalles y el código completo, puedes acceder a este repositorio o echar un vistazo a la documentación de mi Trabajo Fin de Grado.
Referencias
[1] A. Cruz-Roa, A. Basavanhally, F. González, H. Gilmore, M. Feldman, S. Ganesan, N. Shih, J. Tomaszewski, & A. Madabhushi, "Automatic detection of invasive ductal carcinoma in whole slide images with convolutional neural networks," In M.N. Gurcan, & A. Madabhushi,eds., (San Diego, California, USA, 2014), p. 904103. https://doi.org/10.1117/12.2043872.